Frontiers in Oncology
Einführung
Glioblastom und anaplastisches Astrozytom (ANA) sind zwei der häufigsten primären Hirntumoren bei Erwachsenen (1). Die Inzidenzraten nehmen zu, insbesondere bei älteren Menschen (1-3). In der klinischen Praxis ist es schwierig, Patienten mit Glioblastom vor der Operation oder Biopsie von denen mit ANA zu unterscheiden, da die Symptome und Anzeichen der beiden Tumoren relativ einheitlich und unspezifisch sind (4, 5). Die Behandlung ist jedoch unterschiedlich, z. B. in Bezug auf das Chemotherapieprotokoll, die Dosierung und die Art der Verabreichung (6). So wird beispielsweise bei Patienten mit ANA (WHO-Grad III) eine Strahlentherapie oder TMZ nach Resektion oder Biopsie empfohlen, während bei Patienten, bei denen ein Glioblastom (WHO-Grad IV) neu diagnostiziert wurde, eine Strahlentherapie plus gleichzeitige TMZ-Therapie, gefolgt von einer adjuvanten TMZ-Therapie, durchgeführt wird. Früheren Studien zufolge wachsen Glioblastom und ANA durch Invasion in normales Hirngewebe, breiten sich über den Liquor aus und reichen über eine einzige Karotis- oder Vertebralarterie hinaus, so dass sie beide schlecht auf eine medizinische Behandlung ansprechen und zu den häufigsten krebsbedingten Todesursachen bei Erwachsenen gehören (7, 8). Außerdem sind die Prognosen von Glioblastom und ANA unterschiedlich. In der älteren Bevölkerung besteht kein signifikanter Unterschied in der Prognose zwischen Glioblastom und ANA, aber in der jüngeren Bevölkerung kann es einen Unterschied geben (9). Daher ist die Unterscheidung zwischen Glioblastom und ANA schwierig, aber entscheidend.
Die Magnetresonanztomographie (MRT) ist aufgrund ihrer multiplanaren Fähigkeit und ihres hervorragenden Weichteilkontrasts das optimale Neuroimaging für die präoperative Diagnose von Glioblastom und ANA. Obwohl einige Studien gezeigt haben, dass das Vorhandensein von ringförmiger Anreicherung und Nekrose auf ein Glioblastom hindeutet, erscheinen sowohl Glioblastom als auch ANA in den meisten Fällen als unregelmäßige Formen auf MR-Bildern (hyperdicht auf der T2-gewichteten Sequenz und hypodens auf der T1-gewichteten Sequenz) mit unterschiedlichem Grad an Gd-basierter Kontrastanreicherung und Ödem, wobei die Unterschiede für das menschliche Auge in der Regel nicht wahrnehmbar waren (10-12).
In jüngster Zeit wurde die Texturanalyse (TA), die auch als Radiomik bekannt ist, in verschiedenen Bereichen eingesetzt. Forscher fanden heraus, dass die TA eine praktikable und vielversprechende Methode zur Erleichterung der Differentialdiagnose ist, da sie die Erfassung zusätzlicher quantitativer Informationen aus MR-Bildern ermöglicht, die für die menschliche Beurteilung unsichtbar sind (13-15). TA beschreibt die Häufigkeitsverteilung und die räumliche Organisation der Voxelwerte, um mögliche Unterschiede im Tumorgewebe aufzudecken (16). In früheren Studien wurde die Durchführbarkeit der Anwendung von TA bei der Differentialdiagnose, der Klassifizierung von Tumorsubtypen und der Erkennung der Heterogenität von Tumorgewebe untersucht (17-19). Über die Anwendung der TA bei der Differenzialdiagnose zwischen Glioblastom und ANA wurde jedoch noch nicht berichtet. Ziel dieser Studie war es, die Unterscheidungsfähigkeit der MRT-Texturanalyse unter Verwendung von Algorithmen des maschinellen Lernens zur Unterscheidung von Glioblastom und ANA zu bewerten.
Materialien und Methoden
Patientenauswahl
Wir haben die Datenbank unserer Einrichtung retrospektiv durchsucht und alle Patienten untersucht, bei denen von Januar 2015 bis Dezember 2018 histopathologisch ein Glioblastom oder ANA diagnostiziert wurde. Die Zulassungskriterien für qualifizierte Patienten waren: (1) schlüssige histopathologische Diagnose von Glioblastom oder ANA; (2) ausführliche elektronische Krankenakten, insbesondere pathologisches Material; (3) diagnostischer MR-Scan in unserer Einrichtung vor der chirurgischen Resektion. Ausschlusskriterien waren: (1) Vorgeschichte einer intrakraniellen Erkrankung (z. B. Hirntrauma, intrakranielle Infektion oder andere Arten von Hirntumoren) unter Berücksichtigung der Beeinträchtigung der Bildintensität durch Narbengewebe; (2) Vorhandensein von Bewegungsartefakten auf der MRT; (3) Vorgeschichte von Behandlungen vor der MRT-Untersuchung (z. B. Operation, Chemo- oder Strahlentherapie); (4) Patienten, die die Kriterien für die Diagnose eines Glioblastoms oder einer ANA gemäß dem WHO-Klassifikationssystem 2016 nicht erfüllten. Ein leitender Neuropathologe mit 10-jähriger Erfahrung beurteilte, ob der Patient die Kriterien (des WHO-Klassifikationssystems 2016) für ein Glioblastom oder eine ANA erfüllte. Der institutionelle Prüfungsausschuss genehmigte diese retrospektive Studie. Die schriftliche Einwilligung der Teilnehmer an dieser Studie wurde eingeholt. Die Ethikkommission der Universität Sichuan und die radiologische Abteilung unserer Einrichtung haben der Verwendung der Statistiken für diese Studie zugestimmt.
MR-Bilderfassung
Für alle in diese Studie eingeschlossenen Patienten standen kontrastverstärkte T1-gewichtete Sequenzen zur Verfügung, die auf einem 3.0T Siemens Trio Scanner mit den folgenden Parametern gewonnen wurden: TR/TE/TI = 1900/2,26/900 ms, Flip-Winkel = 9 °, 20 axiale Schichten mit einer Dicke von = 5 mm, axiales FOV = 25,6 × 25,6 cm2 und Datenmatrix = 256 × 256. Für die kontrastverstärkte T1-gewichtete Bildgebung wurde Gadopentetat-Dimeglumin (0,1 mmol/Kg) als Kontrastmittel verwendet, und die multidirektionalen Daten der kontrastverstärkten MRT wurden während der kontinuierlichen Intervallzeit von 90-250 s gesammelt.
Texturextraktion
In unserer Studie wurde das LifeX-Paket (http://www.lifexsoft.org) verwendet, um Texturmerkmale zu extrahieren. Für die weitere Analyse wurden T1-gewichtete Nachkontrastbilder (T1C) ausgewählt, da sie die Lage und den Rand des Tumors klar darstellen (20). Die Region of Interest (ROI) wurde manuell Schicht für Schicht in der axialen Ebene entlang der Läsionen auf kontrastverstärkten Bildern gezogen, um Texturmerkmale zu erhalten. Zwei erfahrene Neurochirurgen, die die Krankenakten der Patienten und die histopathologische Diagnose nicht kannten, zeichneten die ROI, die anschließend von einem erfahrenen Radiologen und einem erfahrenen Neurochirurgen bearbeitet wurden. Unstimmigkeiten wurden mit dem leitenden Radiologen und dem leitenden Neurochirurgen besprochen und beraten. Insgesamt wurden 40 Texturmerkmale aus den MRT-Bildern extrahiert, darunter minValue, meanValue, maxValue, stdValue und Parameter, die aus sechs Matrizen abgeleitet wurden: Histogramm-basierte Matrix (HISTO), Shape, Gray-Level-Co-Occurrence-Matrix (GLCM), Gray-Level-Run-Length-Matrix (GLRLM), Gray-Level-Zone-Length-Matrix (GLZLM) und Neighborhood-Gray-Level-Dependence-Matrix (NGLDM).
Merkmalsauswahl
Es gab insgesamt 40 Texturmerkmale, die aus sechs ausgewählten Matrizen abgeleitet wurden. Die Erläuterungen zu den 40 Texturmerkmalen finden sich in der ergänzenden Tabelle 1. Die statistischen Angaben zu diesen Texturmerkmalen sind in der ergänzenden Tabelle 2 aufgeführt. Die Merkmalsauswahl wurde durchgeführt, um relevante Merkmale zu bestimmen und dadurch eine Überanpassung zu vermeiden. Außerdem konnte der in dieser Studie verwendete Algorithmus für maschinelles Lernen nicht alle 40 Texturmerkmale in die Analyse einbeziehen. Zur Auswahl der optimalen Texturmerkmale wurden drei unabhängige Methoden verwendet, darunter die Distanzkorrelation, der Least Absolute Shrinkage and Selection Operator (LASSO) und der Gradient Correlation Decision Tree (GBDT). Auf diese Weise wurden drei Untergruppen von Texturmerkmalen gebildet, die drei verschiedene Datensätze darstellten.
Klassifizierung
Die lineare Diskriminanzanalyse (LDA) ist eine robuste Klassifizierungsmethode zur Trennung von zwei Klassen durch die Suche nach der linearen Kombination von Prädiktoren, die die Trennung zwischen den Gruppen maximiert. In dieser Studie wurden drei Klassifikationsmodelle auf der Grundlage des LDA-Algorithmus erstellt: Distanzkorrelation + LDA, LASSO + LDA, und GBDT + LDA. Die Datensätze wurden jeweils unabhängig voneinander in den LDA-Algorithmus eingespeist. Jeder Datensatz wurde nach dem Zufallsprinzip in eine Trainings- und eine Validierungsgruppe im Verhältnis von 4:1 aufgeteilt. Das in der Trainingsgruppe trainierte Modell wurde dann auf die unabhängige Validierungsgruppe angewendet, um seine Leistung zu bewerten. Um die Robustheit des LDA-Algorithmus zu beurteilen, wurde das Verfahren für 100 Zyklen mit unterschiedlichen, zufälligen und unabhängigen Fallzuweisungen wiederholt. Es wurde eine Konfusionsmatrix erstellt, in der die wahre Zuordnung aus der Histopathologie und die Vorhersagen des LDA-Algorithmus verwendet wurden. Die Standardsensitivität, Spezifität, die Flächen unter der Empfängerkennlinie (AUC) und die Genauigkeit wurden sowohl für die Trainings- als auch für die Validierungsgruppe berechnet, um die Unterscheidungsfähigkeit der Modelle zu ermitteln. Der Vergleich von drei Modellen (Distanzkorrelation + LDA, LASSO + LDA und GBDT + LDA) wurde durchgeführt, um das optimale Unterscheidungsmodell für Glioblastom und ANA zu ermitteln. Das Flussdiagramm der MRT-Klassifizierung anhand von Texturmerkmalen ist in Abbildung 1 dargestellt.
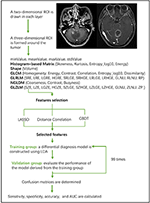
Abbildung 1. Flussdiagramm der MRT-Klassifizierung nach Texturmerkmalen. ANA, anaplastisches Astrozytom; LDA, lineare Diskriminanzanalyse; MRI, Magnetresonanztomographie; LASSO, least absolute shrinkage and selection operator; GBDT, gradient correlation decision tree; HISTO, histogram-based matrix; GLCM, Gray-level co-occurrence matrix; GLRLM, Gray-level run length matrix; GLZLM, Gray-level zone length matrix; NGLDM, Neighborhood gray-level dependence matrix; AUC, area under the receiver operating characteristic curve.
Ergebnisse
Patientenmerkmale
Insgesamt 133 Patienten mit Glioblastom (n = 76) oder ANA (n = 57) erfüllten die Einschlusskriterien. Alle Patienten mit Glioblastom wurden in diese Studie aufgenommen, während 10 Patienten mit ANA gemäß den Ausschlusskriterien ausgeschlossen wurden. Schließlich wurden 76 Patienten mit Glioblastom und 46 Patienten mit ANA in diese Studie aufgenommen. Das Durchschnittsalter der Patienten lag bei 46,9 (15-67) bzw. 40,0 (7-69) Jahren. Alle Patienten wurden in den Jahren 2015 bis 2018 in unserer neurochirurgischen Abteilung einer chirurgischen Tumorresektion unterzogen. Abbildung 2 zeigt zwei Fälle aus der axialen Ebene der kontrastverstärkten Bilder bei Patienten mit Glioblastom und ANA.
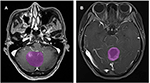
Abbildung 2. Beispiele für zwei Fälle von kontrastverstärkten MR-Bildern bei Patienten mit Glioblastom und ANA. (A) Kontrastverstärkte Bilder mit ANA, (B) kontrastverstärkte Bilder mit Glioblastom. ANA, Anaplastisches Astrozytom.
Glioblastom vs. ANA
In dieser Studie wurden drei Modelle analysiert, darunter Distanzkorrelation + LDA, LASSO + LDA, GBDT + LDA. Die für die Klassifizierung in diesen Modellen verwendeten Texturmerkmale sind in der ergänzenden Tabelle 3 aufgeführt. Die Leistung der einzelnen Modelle wurde in Tabelle 1 dargestellt (einschließlich Sensitivität, Spezifität, Genauigkeit und AUC der Trainings- und Validierungsgruppe). LASSO + LDA erzielte die beste Leistung mit den höchsten AUCs sowohl in der Trainings- als auch in der Validierungsgruppe. Die Sensitivität, Spezifität, Genauigkeit und AUC für die Trainingsgruppe lagen bei 0,989, 0,993, 0,996 bzw. 0,997 und für die Validierungsgruppe bei 0,927, 0,989, 0,968 bzw. 0,974. Darüber hinaus zeigten Distanzkorrelation + LDA und GBDT + LDA ebenfalls eine vielversprechende Fähigkeit zur Unterscheidung von Glioblastom und ANA, mit AUC ≥0,95 sowohl für die Trainings- als auch für die Validierungsgruppen.

Tabelle 1. Unterscheidung zwischen Glioblastom und ANA.
Abbildung 3 zeigt die Beziehung zwischen den kanonischen Unterscheidungsfunktionen aus LASSO + LDA-Modellen für die Glioblastom- und ANA-Gruppen (Dreiecke und Kreise) und für die Gruppenschwerpunkte (Quadrate). In dieser Abbildung wurden nur minimale Überschneidungen festgestellt. Qualitativ konnte die Analyse der durch LASSO ausgewählten Daten Glioblastom von ANA trennen. Abbildung 4 zeigt die Verteilung der direkten LDA-Funktion, die für das Glioblastom und ANA für einen der 100 unabhängigen Trainingszyklen der Datenanalyse ermittelt wurde, um die Leistung des LASSO + LDA-Modells zu veranschaulichen. Es gab deutliche Verschiebungen der LDA-Funktionswerte, mit einer Linksverschiebung für ANA und einer Rechtsverschiebung für Glioblastom.
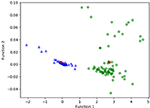
Abbildung 3. Beziehung zwischen den kanonischen Unterscheidungsfunktionen aus LASSO- und LDA-Modellen für die Glioblastom- und ANA-Gruppen (Dreiecke und Kreise) und für die Gruppenschwerpunkte (Quadrate). In dieser Abbildung wurden nur minimale Überschneidungen festgestellt. Qualitativ konnte die Analyse der durch LASSO ausgewählten Daten Glioblastom von ANA trennen. LASSO, least absolute shrinkage and selection operator; LDA, lineare Diskriminanzanalyse; ANA, anaplastisches Astrozytom.
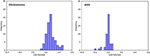
Abbildung 4. Verteilung der für das Glioblastom und ANA ermittelten direkten LDA-Funktion für einen der 100 unabhängigen Trainingszyklen in der Datenanalyse zur Veranschaulichung der Leistung des LASSO + LDA-Modells. Es gab deutliche Verschiebungen der LDA-Funktionswerte, mit einer Linksverschiebung für ANA und einer Rechtsverschiebung für Glioblastom. Zwischen den beiden Gruppen ist eine minimale Überlappung zu beobachten, und zwischen den Diagrammen für Zyklen und Dreiecke ist eine starke qualitative Ähnlichkeit erkennbar. LDA, lineare Diskriminanzanalyse; ANA, anaplastisches Astrozytom; LASSO, kleinste absolute Schrumpfung und Selektionsoperator.
Diskussion
Die Differentialdiagnose zwischen Glioblastom und ANA vor der Behandlung ist wichtig, wenn man den signifikanten Unterschied in der Behandlungsstrategie und der Patientenprognose berücksichtigt. Die MR-Untersuchung, die wichtigste radiologische präoperative Untersuchung bei Hirntumoren, wird wegen ihrer hohen Sensitivität bei der Erkennung von Läsionen dringend empfohlen. Eine genaue Diagnose vor der Operation ist jedoch nach wie vor schwierig, da beide Tumoren auf konventionellen MR-Bildern ähnliche Merkmale aufweisen, die mit dem bloßen Auge nicht zu erkennen sind (4, 5). In dieser Studie haben wir Texturmerkmale extrahiert, die die Bilder quantitativ beschreiben, um die Nutzung der MR-Untersuchung zu maximieren, und drei LDA-basierte Modelle entwickelt. Die Ergebnisse zeigten, dass die MRT-basierte Texturanalyse in Kombination mit dem LDA-Algorithmus eine praktikable Unterscheidung zwischen Glioblastom und ANA ermöglichen kann.
TA ist ein mathematischer Ansatz zur Charakterisierung der Heterogenität von Voxelwerten auf Bildern. Sie kann die räumliche histologische Heterogenität sichtbar machen, die für das menschliche Auge unsichtbar ist (21). Theoretisch könnten die Merkmale von Läsionsbildern aufgrund ihrer unterschiedlichen verstärkten Muster auf MR-Bildern als Texturmerkmale quantitativ analysiert werden (22). Darüber hinaus legen frühere Studien nahe, dass die Texturmerkmale eine Reihe abnormaler pathologischer Prozesse des Tumors wie Ödeme, Ergüsse und Nekrosen widerspiegeln könnten, was einen potenziellen Mechanismus für Texturmerkmale bei der Unterscheidung von Glioblastom und ANA darstellt (23, 24).
Künstliche Intelligenz wurde in der jüngsten Forschung umfassend erforscht. In Kombination mit Texturmerkmalen, die aus Bildern extrahiert werden, soll sie bei der Tumoreinstufung, der klinischen Diagnose und der Ergebnisvorhersage helfen. Eine Studie zur Bewertung der diagnostischen Leistung von TA-basierten Algorithmen des maschinellen Lernens bei der Unterscheidung von PCNSL und Glioblastom ergab eine optimale Leistung mit einer mittleren AUC von 0,921, während die AUC von drei Lesern alle <0,8 waren. Die Forscher kamen daher zu dem Schluss, dass die diagnostische Leistung von TA-basierten Algorithmen des maschinellen Lernens derjenigen von menschlichen Lesern überlegen war (25). Andere Studien mit ähnlicher Zielsetzung zeigten ebenfalls ähnliche Ergebnisse mit AUCs von über 0,85 (18, 26). Darüber hinaus berichteten Forscher, die maschinelles Lernen bei der Klassifizierung von Astrozytomen einsetzen wollten, über eine vielversprechende Fähigkeit zur Unterscheidung (27). In unserer Studie wurden die Klassifizierungsmodelle auf der Grundlage von LDA-Algorithmen erstellt. LDA ist ein statistischer Klassifikator, der die eingegebenen Parameter zu einer Diskriminanzfunktion kombiniert, um die Fälle in verschiedene Gruppen einzuteilen (28). Unsere Ergebnisse zeigten, dass das LDA-basierte Modell eine vielversprechende Leistung bei der genauen Diagnose zwischen Glioblastom und ANA darstellt.
Die Annahme optimaler Merkmale für Algorithmen des maschinellen Lernens war eine Herausforderung, aber im Hinblick auf die diagnostische Leistung notwendig. In früheren Studien wurde die Merkmalsauswahl mit unterschiedlichen Methoden durchgeführt: Mann-Whitney-U-Test mit AUC des ROC, Student’s t-Test mit rekursiver Merkmalseliminierung, Random Forest und entropiebasierte Diskretisierung (18, 25, 29, 30). Anhand der Ergebnisse dieser Studien konnten wir die Schlussfolgerung ziehen, dass die geeignete Auswahlmethode eine Schlüsselrolle für die Leistung des Klassifikators spielt. In unserer Studie wurde eine relativ große Anzahl von Parametern aus verschiedenen Matrizen extrahiert, was die Chance auf die Auswahl der optimalen Merkmale erhöht, aber auch die Schwierigkeit der Auswahl steigert. Daher wurden drei Methoden zur Merkmalsauswahl (Distanzkorrelation, LASSO und GBDT) bewertet, um die Methode mit der besten Leistung auszuwählen. Die Ergebnisse dieser Studie zeigten, dass LASSO+LDA das geeignete Unterscheidungsmodell für Glioblastome von ANA war, mit dem höchsten AUC in der Testgruppe von 0,997. LASSO wurde in einer früheren Studie als nichtlineare Variablenauswahlmethode für neuronale Netze vorgeschlagen, die den Vorteil hat, die gemeinsame Summe der quadratischen Fehler zu minimieren. Es könnte interpretierbare Modelle erzeugen (ähnlich wie die Teilmengenauswahl) und gleichzeitig die Stabilität der Ridge-Regression aufweisen. Eine frühere Studie hat gezeigt, dass diese Methode anderen modernen Methoden der Variablenauswahl überlegen ist (31). Wir müssen die Ergebnisse jedoch vorsichtig interpretieren, da der zusätzliche Informationsgewinn durch den Vergleich verschiedener maschineller Lerntechniken recht begrenzt ist, insbesondere angesichts der Tatsache, dass alle untersuchten Klassifikator-/Merkmalsauswahlmethoden recht vergleichbare Leistungen zu erbringen scheinen und die Varianz in der AUC möglicherweise teilweise auf die statistische Gruppe zurückzuführen ist. Daher kann unsere Studie nur als Hypothesengenerierung für zukünftige, größere Studien betrachtet werden.
Es gab einige Einschränkungen unserer Studie. Erstens war es eine retrospektive Studie eines einzigen Zentrums, was eine Verzerrung bei der Patientenauswahl unvermeidlich machte. Zweitens war die Zahl der eingeschlossenen Patienten relativ gering, und in weiteren Studien wäre eine größere Zahl von Patienten erforderlich, um die Ergebnisse zu validieren. Drittens werden ANA jetzt gemäß der 2016 von der Weltgesundheitsorganisation erstellten Klassifikation von Tumoren des zentralen Nervensystems in drei Kategorien eingeteilt: IDH-mutant, IDH-Wildtyp und NOS (32). Die Fähigkeit des maschinellen Lernens zur Unterscheidung von ANA-Subtypen sollte in zukünftigen Studien untersucht werden. Viertens wurden die maschinellen Lernmodelle in unserer Studie nicht in anderen Datensätzen validiert. Wir haben keine Datensätze anderer Institutionen verwendet, weil die Texturmerkmale unterschiedlich sein könnten, wenn sie aus Bildern extrahiert werden, die mit verschiedenen Scannern oder Protokollen aufgenommen wurden. Dies könnte als ein zweischneidiges Schwert betrachtet werden. Einerseits könnte ein Satz kontrollierter Variablen zur Verfügung gestellt werden, andererseits wäre eine breite Anwendung der Ergebnisse nicht gewährleistet. Das Analyseprotokoll und das Bildverarbeitungsverfahren waren Open-Source-Pakete, und es sind Studien mit einer großen Population erforderlich, um unsere Ergebnisse zu validieren und zu reproduzieren.
Schlussfolgerung
In dieser Arbeit haben wir quantitative Parameter aus kontrastverstärkten MR-Bildern extrahiert und drei Methoden zur Merkmalsauswahl verwendet, um die am besten unterscheidenden Parameter auszuwählen. Anschließend haben wir den LDA-Algorithmus zur Analyse der ausgewählten Parameter angewandt. Unsere Studie zeigt, dass Texturmerkmale eine vielversprechende Fähigkeit zur Unterscheidung von Glioblastomen und ANA aufweisen. Multizentrische Studien mit einer größeren Anzahl von Patienten sind erforderlich, um dieses vorläufige Ergebnis zu bestätigen.
Datenverfügbarkeit
Wir freuen uns, unsere Daten allen qualifizierten Forschern ohne Vorbehalt zur Verfügung zu stellen. Bitte kontaktieren Sie den korrespondierenden Autor, wenn Sie etwas benötigen.
Ethikerklärung
Die Studien mit menschlichen Teilnehmern wurden von der Ethikkommission der Universität Sichuan geprüft und genehmigt. Die schriftliche Einwilligung zur Teilnahme an dieser Studie wurde von den Erziehungsberechtigten/Verwandten der Teilnehmer erteilt. Für die Veröffentlichung der in diesem Artikel enthaltenen potenziell identifizierbaren Bilder oder Daten wurde die schriftliche Einwilligung der Teilnehmer und der Erziehungsberechtigten bzw. der nächsten Angehörigen der Minderjährigen eingeholt.
Beiträge der Autoren
ZT, CC, JX und XM trugen zur Konzeption und Gestaltung der Studie bei. CC und XO rekrutierten die in Frage kommenden Patienten und beschafften die Krankenakten und MRT-Bilder der einzelnen Patienten. YF und ZT führten die Texturanalyse durch. JW erstellte die Modelle und führte weitere statistische Analysen durch. ZT schrieb den ersten Entwurf des Manuskripts. CC schrieb Teile des Manuskripts. Alle Autoren trugen zur Überarbeitung des Manuskripts bei, lasen und genehmigten die eingereichte Version.
Erklärung zu Interessenkonflikten
Die Autoren erklären, dass die Forschung in Abwesenheit jeglicher kommerzieller oder finanzieller Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Ergänzendes Material
Das ergänzende Material zu diesem Artikel finden Sie online unter: https://www.frontiersin.org/articles/10.3389/fonc.2019.00876/full#supplementary-material
Abkürzungen
ANA, Anaplastisches Astrozytom; LDA, Lineare Diskriminanzanalyse; MRI, Magnetresonanztomographie; MR, Magnetresonanz; LASSO, Least absolute shrinkage and selection operator; GBDT, Gradient correlation decision tree; CSF, Cerebrospinal fluid; TA, Texturanalyse; ROI, Regions of interest; HISTO, Histogramm-basierte Matrix; GLCM, Grey-Level-Co-Occurrence-Matrix; GLRLM, Grey-Level-Run-Length-Matrix; GLZLM, Grey-Level-Zone-Length-Matrix; NGLDM, Neighborhood-Grey-Level-Dependence-Matrix; AUC, Fläche unter der Receiver-Operating-Characteristic-Curve; PCNSL, Primäres Zentralnervensystem-Lymphom; MLP, Multilayer-Perceptron; IDH, Isocitratdehydrogenase.
1. Torre LA, Bray F, Siegel RL, Ferlay J, Lortet-Tieulent J, Jemal A. Global cancer statistics, 2012. CA Cancer J Clin. (2015) 65:87-108. doi: 10.3322/caac.21262
PubMed Abstract | CrossRef Full Text | Google Scholar
2. Chen W, Zheng R, Baade PD, Zhang S, Zeng H, Bray F, et al. Cancer statistics in China, 2015. CA Cancer J Clin. (2016) 66:115-32. doi: 10.3322/caac.21338
PubMed Abstract | CrossRef Full Text | Google Scholar
3. Wen PY, Kesari S. Malignant gliomas in adults. N Engl J Med. (2008) 359:492-507. doi: 10.1056/NEJMra0708126
PubMed Abstract | CrossRef Full Text | Google Scholar
4. Grimm SA, Chamberlain CM. Anaplastic astrocytoma. CNS Oncol. (2016) 5:145-57. doi: 10.2217/cns-2016-0002
PubMed Abstract | CrossRef Full Text | Google Scholar
5. Batash R, Asna N, Schaffer P, Francis N, Schaffer M. Glioblastoma multiforme, diagnosis and treatment; recent literature review. Curr Med Chem. (2017) 24:3002-9. doi: 10.2174/0929867324666170516123206
PubMed Abstract | CrossRef Full Text | Google Scholar
6. Weller M, van den Bent M, Hopkins K, Tonn JC, Stupp R, Falini AE, et al. EANO guideline for the diagnosis and treatment of anaplastic gliomas and glioblastoma. Lancet Oncol. (2014) 15:e395-403. doi: 10.1016/s1470-2045(14)70011-7
PubMed Abstract | CrossRef Full Text | Google Scholar
7. Souhami L, Seiferheld W, Brachman D, Podgorsak EB, Werner-Wasik M, Lustig R, et al. Randomisierter Vergleich von stereotaktischer Radiochirurgie gefolgt von konventioneller Strahlentherapie mit Carmustin mit konventioneller Strahlentherapie mit Carmustin bei Patienten mit Glioblastoma multiforme: Bericht über das Protokoll 93-05 der Radiation Therapy Oncology Group. Int J Radiat Oncol Biol Phys. (2004) 60:853-60. doi: 10.1016/j.ijrobp.2004.04.011
PubMed Abstract | CrossRef Full Text | Google Scholar
8. Burger PC, Heinz ER, Shibata T, Kleihues P. Topographische Anatomie und CT-Korrelationen beim unbehandelten Glioblastoma multiforme. J Neurosurg. (1988) 68:698-704. doi: 10.3171/jns.1988.68.5.0698
PubMed Abstract | CrossRef Full Text | Google Scholar
9. Wick W, Platten M, Meisner C, Felsberg J, Tabatabai G, Simon M, et al. Temozolomide chemotherapy alone versus radiotherapy alone for malignant astrocytoma in the elderly: the NOA-08 randomised, phase 3 trial. Lancet Oncol. (2012) 13:707-15. doi: 10.1016/s1470-2045(12)70164-x
PubMed Abstract | CrossRef Full Text | Google Scholar
10. Chen W, Silverman HD. Fortschritte bei der Bewertung von primären Hirntumoren. Semin Nucl Med. (2008) 38:240-50. doi: 10.1053/j.semnuclmed.2008.02.005
PubMed Abstract | CrossRef Full Text | Google Scholar
11. Burger PC, Vogel FS, Green SB, Strike AT. Glioblastoma multiforme und anaplastisches Astrozytom. Pathologische Kriterien und prognostische Implikationen. Cancer. (1985) 56:1106-11.
PubMed Abstract | Google Scholar
12. Omuro A, DeAngelis ML. Glioblastom und andere bösartige Gliome: eine klinische Übersicht. JAMA. (2013) 310:1842-50. doi: 10.1001/jama.2013.280319
PubMed Abstract | CrossRef Full Text | Google Scholar
13. Lakhman Y, Veeraraghavan H, Chaim J, Feier D, Goldman DA, Moskowitz CS, et al. Differentiation of uterine leiomyosarcoma from atypical leiomyoma: diagnostic accuracy of qualitative MR imaging features and feasibility of texture analysis. Eur Radiol. (2017) 27:2903-15. doi: 10.1007/s00330-016-4623-9
PubMed Abstract | CrossRef Full Text | Google Scholar
14. Skogen K, Schulz A, Dormagen JB, Ganeshan B, Helseth E, Server A. Diagnostic performance of texture analysis on MRI in grading cerebral gliomas. Eur J Radiol. (2016) 85:824-9. doi: 10.1016/j.ejrad.2016.01.013
PubMed Abstract | CrossRef Full Text | Google Scholar
15. Gibbs P, Turnbull WL. Textural analysis of contrast-enhanced MR images of the breast. Magn Reson Med. (2003) 50:92-8. doi: 10.1002/mrm.10496
PubMed Abstract | CrossRef Full Text | Google Scholar
16. Castellano G, Bonilha L, Li LM, Cendes F. Texture analysis of medical images. Clin Radiol. (2004) 59:1061-9. doi: 10.1016/j.crad.2004.07.008
PubMed Abstract | CrossRef Full Text | Google Scholar
17. Davnall F, Yip CS, Ljungqvist G, Selmi M, Ng F, Sanghera B, et al. Assessment of tumor heterogeneity: an emerging imaging tool for clinical practice? Insights Imaging. (2012) 3:573-89. doi: 10.1007/s13244-012-0196-6
PubMed Abstract | CrossRef Full Text | Google Scholar
18. Xiao DD, Yan PF, Wang YX, Osman MS, Zhao YH. Glioblastom und primäres Lymphom des Zentralnervensystems: Präoperative Differenzierung mittels MRT-basierter 3D-Texturanalyse. Clin Neurol Neurosurg. (2018) 173:84-90. doi: 10.1016/j.clineuro.2018.08.004
PubMed Abstract | CrossRef Full Text | Google Scholar
19. Itakura H, Achrol AS, Mitchell LA, Loya JJ, Liu T, Westbroek EM, et al. Magnetresonanzbildmerkmale identifizieren phänotypische Glioblastom-Subtypen mit unterschiedlichen molekularen Signalwegen. Sci Transl Med. (2015) 7:303ra138. doi: 10.1126/scitranslmed.aaa7582
PubMed Abstract | CrossRef Full Text | Google Scholar
20. Nioche C, Orlhac F, Boughdad S, Reuze S, Goya-Outi J, Robert C, et al. LIFEx: a freeware for radiomic feature calculation in multimodality imaging to accelerate advances in the characterization of tumor heterogeneity. Cancer Res. (2018) 78:4786-9. doi: 10.1158/0008-5472.Can-18-0125
PubMed Abstract | CrossRef Full Text | Google Scholar
21. Hu LS, Ning S, Eschbacher JM, Gaw N, Dueck AC, Smith KA, et al. Multi-parametric MRI and texture analysis to visualize spatial histologic heterogeneity and tumor extent in glioblastoma. PLoS ONE. (2015) 10:e0141506. doi: 10.1371/journal.pone.0141506
PubMed Abstract | CrossRef Full Text | Google Scholar
22. Florez E, Nichols T, Parker EE, Lirertte ST, Howard CM, Fatemi A. Multiparametric magnetic resonance imaging in the assessment of primary brain tumors through radiomic features: a metric for guided radiation treatment planning. Cureus. (2018) 10:e3426. doi: 10.7759/cureus.3426
PubMed Abstract | CrossRef Full Text | Google Scholar
23. Mahmoud-Ghoneim D, Alkaabi MK, de Certaines JD, Goettsche MF. Der Einfluss des Bilddynamikbereichs auf die Texturklassifizierung der weißen Substanz des Gehirns. BMC Med Imaging. (2008) 8:18. doi: 10.1186/1471-2342-8-18
PubMed Abstract | CrossRef Full Text | Google Scholar
24. Ahn SY, Park CM, Park SJ, Kim HJ, Song C, Lee SM, et al. Prognostic value of computed tomography texture features in non-small cell lung cancers treated with definitive concomitant chemoradiotherapy. Invest Radiol. (2015) 50:719-25. doi: 10.1097/rli.0000000000000174
PubMed Abstract | CrossRef Full Text | Google Scholar
25. Suh HB, Choi YS, Bae S, Ahn SS, Chang JH, Kang SG, et al. Primary central nervous system lymphoma and atypical glioblastoma: Differentiation using radiomics approach. Eur Radiol. (2018) 28:3832-9. doi: 10.1007/s00330-018-5368-4
PubMed Abstract | CrossRef Full Text | Google Scholar
26. Mouthuy N, Cosnard G, Abarca-Quinones J, Michoux N. Multiparametric magnetic resonance imaging to differentiate high-grade gliomas and brain metastases. J Neuroradiol. (2012) 39:301-7. doi: 10.1016/j.neurad.2011.11.002
PubMed Abstract | CrossRef Full Text | Google Scholar
27. Glotsos D, Spyridonos P, Cavouras D, Ravazoula P, Dadioti PA, Nikiforidis G. An image-analysis system based on support vector machines for automatic grade diagnosis of brain-tumour astrocytomas in clinical routine. Med Inform Internet Med. (2005) 30:179-93. doi: 10.1080/14639230500077444
PubMed Abstract | CrossRef Full Text | Google Scholar
28. Dellacasa Bellingegni A, Gruppioni E, Colazzo G, Davalli A, Sacchetti R, Guglielmelli E, et al. NLR, MLP, SVM, and LDA: a comparative analysis on EMG data from people with trans-radial amputation. J Neuroeng Rehabil. (2017) 14:82. doi: 10.1186/s12984-017-0290-6
PubMed Abstract | CrossRef Full Text | Google Scholar
29. Ion-Margineanu A, Van Cauter S, Sima DM, Maes F, Sunaert S, Himmelreich U, et al. Classifying glioblastoma multiforme follow-up progressive vs. responsive forms using multi-parametric MRI features. Front Neurosci. (2016) 10:615. doi: 10.3389/fnins.2016.00615
PubMed Abstract | CrossRef Full Text | Google Scholar
30. Fetit AE, Novak J, Peet AC, Arvanitits NT. Dreidimensionale Texturmerkmale der konventionellen MRT verbessern die diagnostische Klassifikation von Hirntumoren bei Kindern. NMR Biomed. (2015) 28:1174-84. doi: 10.1002/nbm.3353
PubMed Abstract | CrossRef Full Text | Google Scholar
31. Sun K, Huang SH, Wong DS, Jang SS. Design und Anwendung einer Variablenauswahlmethode für ein mehrschichtiges Perceptron-Neuronalnetz mit LASSO. IEEE Trans Neural Netw Learn Syst. (2017) 28:1386-96. doi: 10.1109/tnnls.2016.2542866
PubMed Abstract | CrossRef Full Text | Google Scholar
32. Louis DN, Perry A, Reifenberger G, von Deimling A, Figarella-Branger D, Cavenee WK, et al. The 2016 World Health Organization classification of tumors of the central nervous system: a summary. Acta Neuropathol. (2016) 131:803-20. doi: 10.1007/s00401-016-1545-1
PubMed Abstract | CrossRef Full Text | Google Scholar




